Cientistas estão criando proteínas artificiais para o nosso corpo
Nosso corpo produz cerca de 20 mil tipos diferentes de proteínas, do colágeno em nossa pele até a hemoglobina em nosso sangue. Algumas tomam a forma de folhas moleculares. Outras são esculpidas em fibras, caixas, túneis, até mesmo tesouras.
A forma específica de uma proteína permite que ele faça um trabalho específico, seja transportar oxigênio pelo corpo ou ajudar a digerir alimentos.
Os cientistas têm estudado as proteínas há quase dois séculos e, ao longo desse tempo, descobriram como as células as criam a partir de blocos simples. Há muito tempo eles sonham em remontar esses elementos, criando novas proteínas não encontradas na natureza.
Mas havia um grande mistério: como os blocos de uma proteína tomam sua forma final. David Baker, de 55 anos, diretor do Instituto de Design de Proteínas da Universidade de Washington, investiga esse enigma há 25 anos.
Agora, parece que ele e seus colegas o solucionaram. Graças, em parte, aos computadores conectados e aos celulares de mais de um milhão de voluntários, os cientistas descobriram como escolher os blocos necessários para criar uma proteína que assumirá o formato que desejarem.
Em uma série de artigos publicados este ano, Baker e seus colegas revelaram os resultados deste trabalho. Eles produziram milhares de diferentes tipos de proteínas, que assumem a forma que os cientistas previram. Muitas vezes, são profundamente diferentes de qualquer outra encontrada natureza.
Essa nova habilidade levou a um profundo avanço científico: proteínas celulares projetadas pelo homem e não pela natureza. "Agora podemos construir proteínas do zero para fazer o que queremos", disse Baker.
Ferramentas moleculares
Logo, os cientistas serão capazes de construir ferramentas moleculares precisas para uma vasta gama de tarefas, ele prevê. Até agora, sua equipe criou proteínas para fins que vão desde lutar contra vírus da gripe até quebrar o glúten em alimentos e detectar vestígios de drogas opioides.
William DeGrado, biólogo molecular da Universidade da Califórnia, em San Francisco, afirmou que os estudos recentes de Baker e seus colegas representam um marco nesta linha de pesquisa científica. "Na década de 1980, sonhávamos em ter resultados tão impressionantes", disse ele.
Toda proteína na natureza é codificada por um gene. Com essa sequência de DNA como guia, uma célula monta uma proteína correspondente com blocos conhecidos como aminoácidos.
Selecionando entre cerca de 20 tipos diferentes, a célula constrói uma cadeia de aminoácidos. Essa cadeia pode se estender por dezenas, centenas ou mesmo milhares de unidades de comprimento. Uma vez que a célula termina, a corrente se dobra em si, geralmente em apenas alguns centésimos de segundo.
Proteínas dobram porque cada aminoácido possui uma carga elétrica. Partes da cadeia de proteínas atraem umas às outras enquanto outras partes são repelidas. Algumas ligações entre os aminoácidos se rendem facilmente à essas forças; os laços rígidos resistem.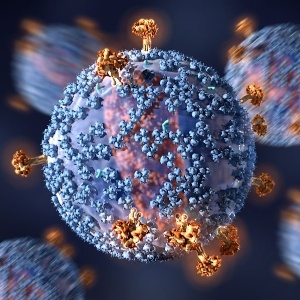
A combinação de todas essas forças atômicas torna cada proteína um quebra-cabeça molecular surpreendente. Quando Baker fez sua pós-graduação na Universidade da Califórnia, em Berkeley, ninguém sabia como prever a forma em que uma cadeia de aminoácidos iria se dobrar. Os cientistas de proteínas se referiam ao enigma simplesmente como "o problema da dobra".
O problema da dobra deixou os pesquisadores na pré-história em termos da manipulação desses importantes elementos biológicos. Eles só podiam usar proteínas que encontrassem na natureza, como os primeiros seres humanos que procuravam rochas afiadas para separar a carne dos ossos.
Usamos proteínas há milhares de anos. Os primeiros fabricantes de queijos, por exemplo, faziam o leite coalhar adicionando um pedaço de estômago de bezerro a ele. A proteína de quimosina, produzida no estômago, transformava o leite líquido em uma forma semissólida.
Hoje, os cientistas ainda procuram maneiras de aproveitar as proteínas. Alguns pesquisadores as estudam em conchas de mariscos na esperança de criar uma armadura corporal mais forte, por exemplo. Outros estão investigando a seda de aranha para fazer cordões de paraquedas. Os pesquisadores também experimentam mudanças modestas nas proteínas naturais para ver se os ajustes permitem fazer coisas novas.
Para Baker e muitos outros cientistas de proteínas, no entanto, esse tipo de manipulação tem sido profundamente insatisfatório. As proteínas encontradas na natureza representam apenas uma fração minúscula do "universo proteico" – todas as proteínas que poderiam ser feitas com diferentes combinações de aminoácidos.
"Quando as pessoas querem uma nova proteína, buscam na natureza coisas que já existem. Não há criação envolvida", disse ele.
Baker tem uma aparência irreverente e uma atitude alegre – mas isso esconde um impulso implacável.
Depois de se formar em Berkeley e se mudar para a Universidade de Washington, juntou-se à tentativa para resolver o problema da dobra. Ele e seus colegas aproveitaram o fato de que as proteínas naturais são um tanto parecidas entre si.
Novas proteínas não surgem do nada; todas evoluem a partir de proteínas ancestrais. Sempre que os cientistas descobriam a forma de uma determinada proteína, também eram capazes de fazer suposições com base em informações sobre os formatos de proteínas relacionadas.
Os cientistas também se basearam no fato de que muitas proteínas são feitas de partes parecidas. Uma característica comum é um trecho espiral de aminoácidos chamado alfa-hélice. Os pesquisadores aprenderam a reconhecer a série de aminoácidos que se dobram nessas espirais.
No final da década de 1990, a equipe da Universidade de Washington passou a usar softwares de estudos individuais de proteínas complexas. O laboratório decidiu criar uma linguagem comum para todo esse código. Assim, os pesquisadores poderiam acessar o conhecimento coletivo sobre proteínas.
Em 1998, lançaram uma plataforma chamada Rosetta, que os cientistas usam para construir cadeias virtuais de aminoácidos e, em seguida, calculam a forma mais provável em que elas irão se dobrar.
Uma comunidade de cientistas de proteínas, conhecida como Rosetta Commons, se formou em torno da plataforma. Nos últimos 20 anos, eles têm melhorado o software e o usam para entender melhor as proteínas – como suas formas permitem seu funcionamento.
Em 2005, Baker lançou um programa chamado Rosetta@home, que recrutou voluntários para doar tempo de processamento em seus computadores domésticos e, com o tempo, nos telefones Android. Nos últimos 12 anos, 1.266.542 pessoas se juntaram à comunidade Rosetta@home.
Passo a passo, a Rosetta ficou mais poderosa e sofisticada, e os cientistas conseguiram usar o poder de processamento comunitário para simular proteínas dobráveis mais detalhadamente. As previsões ficaram surpreendentemente mais precisas.
Fiat lux
Os pesquisadores foram além das proteínas que já existem para proteínas com sequências não naturais. Para ver como essas proteínas não naturais pareciam na vida real, os cientistas sintetizaram genes para elas e as conectaram a células de levedura, que então produziam as criações do laboratório.
"Há sutilezas nas proteínas que ocorrem naturalmente e que ainda não entendemos. Mas resolvemos a maior parte do problema da dobra", disse Baker.
Esses avanços deram à equipe de Baker a confiança para enfrentar um desafio ainda maior: começaram a projetar proteínas do zero para trabalhos específicos. Os pesquisadores partiam de uma tarefa que queriam que a proteína fizesse, e depois descobriam a série de aminoácidos que se dobraria do jeito certo para fazer o trabalho.
Em uma de suas experiências, eles se juntaram a Ian Wilson, virologista do Instituto de Pesquisa Scripps, para desenvolver uma proteína de combate à gripe.
Fim dos vírus?
Wilson tem procurado formas de neutralizar a infecção, e seu laboratório identificou um alvo particularmente promissor: um bolso na superfície do vírus. Se os cientistas pudessem criar uma proteína que se encaixasse nesse bolso, isso poderia impedir o vírus de entrar nas células.
A equipe de Baker usou a Rosetta para projetar tal proteína, apontando sua busca para vários milhares de cadeias de aminoácidos que poderiam fazer o trabalho. Eles simularam a dobra de cada uma, procurando as combinações que poderiam caber na fenda viral.
Os pesquisadores então usaram a levedura artificial para transformar os semifinalistas em proteínas reais. Jogaram as proteínas nos vírus da gripe. Algumas grudaram nos vírus melhor do que outras, e as criações moleculares foram refinadas até chegar à chamada HB1.6928.2.3.
Para ver a eficácia da HB1.6928.2.3 em interromper infecções por gripe, eles realizaram experimentos em camundongos. As proteínas foram pulverizadas no nariz dos camundongos, que depois receberam fortes doses de influenza, o que normalmente seria fatal.
Mas a proteína forneceu 100% de proteção. Resta saber se a HB1.6928.2.3 pode provar sua eficácia em teste com humanos. "Seria bom ter uma droga na linha de frente se uma nova pandemia estivesse prestes a acontecer", disse Wilson.
A HB1.6928.2.3 é apenas uma das várias proteínas que Baker e seus colegas desenvolveram e testaram. Eles também produziram uma molécula que bloqueia a toxina do botulismo e uma que pode detectar pequenas quantidades do opioide fentanil. Outra proteína pode ajudar os celíacos quebrando as moléculas de glúten nos alimentos.
Na semana passada, a equipe de Baker apresentou um dos seus projetos mais ambiciosos: uma concha proteica que pode transportar genes.
Os pesquisadores projetaram proteínas que se juntam como peças de lego, se agrupando em uma esfera oca. No processo, também podem incluir genes e transportar a carga com segurança por horas na corrente sanguínea de camundongos.
Essas conchas têm semelhanças impressionantes com os vírus, embora faltem os meios moleculares para invadir células. "Às vezes, nós as chamamos de não-vírus", disse Baker.
Para reverter doenças hereditárias
Vários pesquisadores estão fazendo experimentos com vírus como meio para distribuir genes pelo corpo. Esses genes podem reverter distúrbios hereditários; em outros experimentos, eles se mostram promissores como uma forma de reprogramar células imunes para combater o câncer.
Mas, como produto de bilhões de anos de evolução, os vírus muitas vezes não funcionam bem como transportadores de genes. "Se construímos um sistema de distribuição desde o início, isso deve funcionar melhor", disse Baker.
Gary Nabel, diretor científico da farmacêutica Sanofi, disse que a nova pesquisa pode levar à invenção de moléculas ainda inimagináveis. "É um novo território, porque você não está moldando proteínas existentes", disse ele.
Por enquanto, Baker e seus colegas só podem fazer proteínas de cadeia curta. Isso se deve, em parte, ao custo envolvido na fabricação de trechos de DNA para codificar proteínas.
Mas essa tecnologia está se desenvolvendo tão rapidamente que a equipe agora testa proteínas maiores e mais longas que podem desempenhar funções mais complexas – entre elas, lutar contra o câncer.
Na imunoterapia contra o câncer, o sistema imunológico reconhece as células cancerígenas pelas proteínas distintas em sua superfície. O sistema imunológico depende de anticorpos que possam reconhecer apenas uma única proteína.
Baker quer criar proteínas que desencadeiem uma resposta somente depois que bloquearem vários tipos de proteínas na superfície de células cancerosas ao mesmo tempo. Ele suspeita que essas moléculas serão mais capazes de reconhecer as células cancerosas, sem mexer nas saudáveis.
Ele disse que, essencialmente, estão criando moléculas que podem fazer cálculos de lógica simples. De fato, espera acabar criando máquinas moleculares.
Nossas células geram combustível com algo como um motor, uma proteína gigantesca chamada ATP sintase, que atua como um tipo de roda d'água. À medida que os prótons carregados positivamente são despejados por um anel de aminoácidos, ela gira cem vezes por segundo. A ATP sintase aproveita essa energia para construir uma molécula de combustível chamada ATP.
Deverá ser possível construir outras máquinas moleculares tão complexas conforme os cientistas aprenderem mais sobre como as grandes proteínas se formam, disse Baker.
"Há muitas coisas que surgiram na natureza apenas por acaso. Conforme entendemos cada vez mais os princípios básicos, devemos ser capazes de fazer muito melhor."






ID: {{comments.info.id}}
URL: {{comments.info.url}}
Ocorreu um erro ao carregar os comentários.
Por favor, tente novamente mais tarde.
{{comments.total}} Comentário
{{comments.total}} Comentários
Seja o primeiro a comentar
Essa discussão está encerrada
Não é possivel enviar novos comentários.
Essa área é exclusiva para você, assinante, ler e comentar.
Só assinantes do UOL podem comentar
Ainda não é assinante? Assine já.
Se você já é assinante do UOL, faça seu login.
O autor da mensagem, e não o UOL, é o responsável pelo comentário. Reserve um tempo para ler as Regras de Uso para comentários.